- 1.24 MB
- 2022-08-30 发布
- 1、本文档由用户上传,淘文库整理发布,可阅读全部内容。
- 2、本文档内容版权归属内容提供方,所产生的收益全部归内容提供方所有。如果您对本文有版权争议,请立即联系网站客服。
- 3、本文档由用户上传,本站不保证质量和数量令人满意,可能有诸多瑕疵,付费之前,请仔细阅读内容确认后进行付费下载。
- 网站客服QQ:403074932
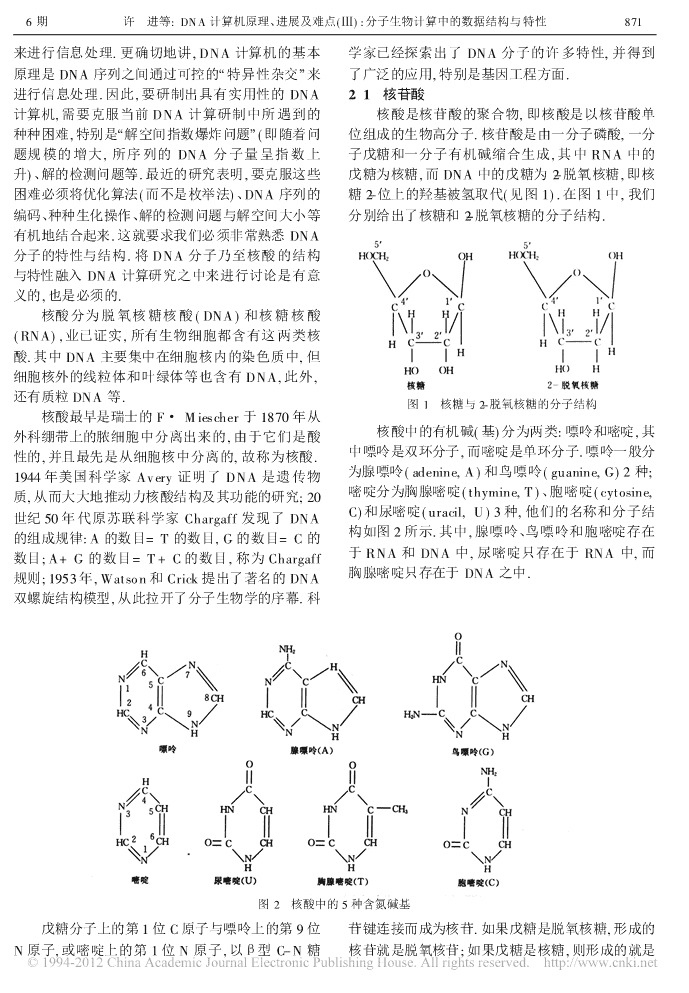
第30卷第6期计算机学报Vol.30No.62007年6月CHINESEJOURNALOFCOMPUTERSJune2007DNA计算机原理、进展及难点(Ⅲ):分子生物计算中的数据结构与特性1),2)2)2)3)许进张社民范月科郭养安1)(北京大学信息科学技术学院北京100871)2)(华中科技大学分子生物计算机研究所武汉430074)3)(西安老年大学西安710014)摘要分子生物计算是指以生物大分子作为数据来进行信息处理的计算模式.目前的分子生物计算主要包含DNA计算、RNA计算和蛋白质计算这三种计算模型.另外,还有一些学者提出采用PNA分子进行计算.但由于PNA计算、RNA计算和蛋白质计算目前还没有一些实质性的突破,故在此不做讨论.研究掌握作为数据的DNA分子特性与结构,显然是DNA计算中的一个基本问题.因而文中主要对各种DNA分子的结构与特征进行讨论.针对问题的不同,模型的不同,采用的DNA分子类型也不同,目前主要用到的是单链的、双链的和具有粘性末端的DNA分子.其次用到的是发夹构型的DNA分子、质粒DNA分子等.文中特别讨论了作为数据的DNA分子与相应的生物计算模型有机相结合的一些基本的问题.关键词DNA计算;DNA分子的结构与类型中图法分类号TP301DNAComputerPrinciple,AdvancesandDifficulties(Ⅲ):TheStructureandCharacterof"Data"inDNAComputing1),2)2)2)3)XUJinZHANGShe-MinFANYue-KeGUOYang-An1)(SchoolofElectronicEngineeringandComputerScience,PekingUniversity,Beijing100871)2)(InstituteofBiomolecularComputer,HuazhongUniversityofScienceandTechnology,Wuhan430074)3)(XicanAgednessUniversity,Xican710014)AbstractBiomolecularcomputing,includingDNAcomputing,RNAcomputingandproteincomputing,isakindofcomputingmodeofinformationprocessingwhose"data"isbiomolecule.Inaddition,thebiomolecularcomputingmodeusingPNAmoleculeas"data"waspresented.ThispaperdoesnotdiscussPNAcomputing,RNAcomputingandproteincomputingbecausetheirresearchresultsareless.Clearly,itisafoundationproblemtoknowwellthestructureandcharacterofthe"data"(thatisDNAmolecules)inDNAcomputing.So,thispapermainlydis-cussesthestructureandcharactersofseveraltypeofDNAmolecule.WeusuallyadoptdifferentDNAmoleculefordifferentproblemormodels.Now,themainkindsofDNAmoleculesusedinDNAcomputingaresingle,doubleandstickerterminalDNAmolecules.Secondly,theDNAmo-leculesusedinDNAcomputinghavealsohairpinDNAmoleculesandplasmidDNAmolecule.Espe-cially,therelationshipbetweenthe"data"(DNAmolecule)andcorrespondingbiomolecularmodelisdisscuss.KeywordsDNAcomputing;thestructureandtypesofDNAmolecules收稿日期:2007-03-24.本课题得到国家自然科学基金重点、面上资助项目(60533010,30670540)和国家/八六三0高技术研究发展计划项目基金(2006AA01Z104)资助.许进,男,1959年生,教授,博士生导师,主要研究领域为生物计算机、图论与系统工程、生物信息学等.E-mail:jxu@pku.edu.cn;jxu@mail.hust.edu.cn.张社民,男,1962年生,博士研究生,教授,主要研究领域为生物计算机与图论.范月科,男,1959年生,博士研究生,副教授,主要研究方向为DNA计算机、图论等.郭养安,男,1960年生,副教授,主要研究方向为计算机科学与管理科学等.\n870计算机学报2007年问题是一个困难的NP-完全问题,并首次将遗传[12]1引言算法应用于DNA计算中的编码搜索和优化;[13]Frutos等提出了模板编码的思想;Hartemink等分子生物计算主要分为三种类型:蛋白质计算、建立了一个基于热力学DNA杂交反应的模拟软[14]RNA计算和DNA计算.蛋白质计算模型的研究始件;Deaton首次将信息论中熵的概念引入DNA[15]于20世纪80年代中期,Conrad首先提出了利用蛋计算;Garzon则提出了一个基于4个编码字母的[1]白质作为计算器件的生物计算机模型.1995年,超立方体的概念,试图借鉴二进制超立方体的理论Birge应用细菌视紫红质蛋白分子具有的良好的/二对编码进行研究.2005年,Tanaka等人给出了设计[2]态性0,拟设计、制造一种蛋白质计算机.Birge的具有统一解链温度的多重核酸序列的一种新的算同事,Syracuse大学的其他研究人员应用原型蛋白法,并且在基于最小自由能量的情况下,不和非特定[16]质制备出一种光电器件,它存储信息的能力比目前的物质进行杂交.的电子计算机的存储器高300倍,这种器件含细菌我们课题组最近几年来也对DNA计算编码问[17]视紫红质蛋白,利用激光束进行信息写入和读题进行了研究,得到了基于遗传算法与常规算[3][18]取.但蛋白质计算机均是拟利用蛋白质的二态性法的一些结果,特别是对DNA计算机的编码问来研制模拟图灵机意义下的计算机模型,应属于纳题进行了较为系统的研究.通过近几年的研究,我们米计算机/家族0的一员.而RNA计算与DNA计算逐渐地认识到,DNA计算的编码可分为两种类型,是利用生化反应,更确切地讲,是以核酸分子间的特一类是所谓的基本型编码,另一类是所谓的综合型异性杂交为机理的、不同于蛋白质计算机机理的计编码.我们对这两类编码进行了较为详细、系统的研算模型.由于RNA分子不仅在实验操作上没有究,成果另文讨论.DNA分子容易,而且在分子结构上也不如DNA分DNA计算中的检测问题是当前DNA计算机子处理信息方便,故目前RNA计算的研究相对较研究中最困难的问题之一.目前在DNA计算研究少.有兴趣的读者可参见文献[4-6].中,关于检测问题一直是困惑DNA计算发展的一相对而言,由于Adleman所建立的基于生化反个障碍,基本上是采用常规的生物检测方法,这些方[7]应机理的DNA计算模型在运行速度和信息的存法主要有电泳技术、毛细管电泳技术、PCR扩增方TM储上业已引起许多不同学科的学者们的关注与兴法、层析技术、磁珠分离、Acrydite技术、DNA传趣,使得关于DNA计算的研究发展很快.目前人们感技术、荧光标记方法、分子信标方法、测序技术以在诸如模型、编码、运算系统、解的检测、生物操作技及生物酶技术等.在已有的研究中,大多是采用这些术以及应用等很多方面都有了深入的研究.关于检测技术来寻找问题的解.我们最近的研究结果表DNA计算的模型,从DNA分子特性与生物酶的功明:将DNA计算解的检测问题与诸如编码问题、能机理角度来分,可分为粘贴模型、剪接模型、自动/解空间大小0等问题结合起来综合处理,并将诸如机模型、自组装模型、发夹DNA计算模型、质粒生物酶、DNA分子的发夹构型以及荧光标记,特别DNA计算模型、k-臂DNA计算模型以及RNA计是分子信标技术有机地结合起来,是当前DNA计算模型等;从生化操作角度来分,可分为试管型、表算机研究中关于解的检测问题的一条较好的解决面计算模型、芯片计算模型以及凝胶电泳计算模型途径.以及PCR-DNA计算机模型等;从应用方面看,有基因表达分析的DNA计算模型;从模拟图灵机方面2DNA计算机的数据)))看,已有图灵机-DNA计算机模型、布尔DNA计算核酸的基本结构与基本性质机模型(或称为分子纳米计算机模型)和有穷自动机[8]模型等.编码问题是DNA计算机研究中的一个DNA计算是以DNA分子作为数据,以生物酶关键问题,在此方面,Baum等人首先注意到编码的和生化操作作为信息处理/工具0的一种新的计算模重要性,并给出了降低DNA序列间的相似度的算式.众所周知,电子计算机是依靠电位的高低产生[9]法;随后,Feldkamp等也研究了降低DNA序列/二态0,其中一种状态用0来表示,另一种状态用1[10]间相似度的算法;Garzon给出了DNA计算编码来表示,由此产生0-1序列来进行信息处理;而[11]问题定义;Deaton等人认为DNA计算中的编码DNA计算则是对DNA序列通过可控的生化反应\n6期许进等:DNA计算机原理、进展及难点(Ⅲ):分子生物计算中的数据结构与特性871来进行信息处理.更确切地讲,DNA计算机的基本学家已经探索出了DNA分子的许多特性,并得到原理是DNA序列之间通过可控的/特异性杂交0来了广泛的应用,特别是基因工程方面.进行信息处理.因此,要研制出具有实用性的DNA211核苷酸计算机,需要克服当前DNA计算研制中所遇到的核酸是核苷酸的聚合物,即核酸是以核苷酸单种种困难,特别是/解空间指数爆炸问题0(即随着问位组成的生物高分子.核苷酸是由一分子磷酸,一分题规模的增大,所序列的DNA分子量呈指数上子戊糖和一分子有机碱缩合生成,其中RNA中的升)、解的检测问题等.最近的研究表明,要克服这些戊糖为核糖,而DNA中的戊糖为2-脱氧核糖,即核困难必须将优化算法(而不是枚举法)、DNA序列的糖2-位上的羟基被氢取代(见图1).在图1中,我们编码、种种生化操作、解的检测问题与解空间大小等分别给出了核糖和2-脱氧核糖的分子结构.有机地结合起来.这就要求我们必须非常熟悉DNA分子的特性与结构.将DNA分子乃至核酸的结构与特性融入DNA计算研究之中来进行讨论是有意义的,也是必须的.核酸分为脱氧核糖核酸(DNA)和核糖核酸(RNA),业已证实,所有生物细胞都含有这两类核酸.其中DNA主要集中在细胞核内的染色质中,但细胞核外的线粒体和叶绿体等也含有DNA,此外,还有质粒DNA等.图1核糖与2-脱氧核糖的分子结构核酸最早是瑞士的F#Miescher于1870年从核酸中的有机碱(基)分为两类:嘌呤和嘧啶,其外科绷带上的脓细胞中分离出来的,由于它们是酸中嘌呤是双环分子,而嘧啶是单环分子.嘌呤一般分性的,并且最先是从细胞核中分离的,故称为核酸.为腺嘌呤(adenine,A)和鸟嘌呤(guanine,G)2种;1944年美国科学家Avery证明了DNA是遗传物嘧啶分为胸腺嘧啶(thymine,T)、胞嘧啶(cytosine,质,从而大大地推动力核酸结构及其功能的研究;20C)和尿嘧啶(uracil,U)3种,他们的名称和分子结世纪50年代原苏联科学家Chargaff发现了DNA构如图2所示.其中,腺嘌呤、鸟嘌呤和胞嘧啶存在的组成规律:A的数目=T的数目,G的数目=C的于RNA和DNA中,尿嘧啶只存在于RNA中,而数目;A+G的数目=T+C的数目,称为Chargaff胸腺嘧啶只存在于DNA之中.规则;1953年,Watson和Crick提出了著名的DNA双螺旋结构模型,从此拉开了分子生物学的序幕.科图2核酸中的5种含氮碱基戊糖分子上的第1位C原子与嘌呤上的第9位苷键连接而成为核苷.如果戊糖是脱氧核糖,形成的N原子,或嘧啶上的第1位N原子,以B型C-N糖核苷就是脱氧核苷;如果戊糖是核糖,则形成的就是\n872计算机学报2007年核糖核苷.核糖核苷共有4个:腺嘌呤核苷(腺苷)、(脱氧腺苷)、鸟嘌呤脱氧核苷(脱氧鸟苷)、胸腺嘧啶鸟嘌呤核苷(鸟苷)、尿嘧啶核苷(尿苷)、胞嘧啶核苷脱氧核苷(胸苷)和胞嘧啶脱氧核苷(脱氧胞苷).如(胞苷);脱氧核苷有4个,它们是:腺嘌呤脱氧核苷图3给出了全部4个核糖核苷和全部4脱氧核苷.图34个核糖核苷和4个脱氧核苷核苷分子中的戊糖环上的羟基磷酸化,形成核核糖或脱氧核糖结合的部位通常是核糖或脱氧核糖苷酸,有时也称为磷酸核苷.从结构上讲,核苷与磷的第3c位或第5c位碳原子.如图4给出了5c-腺嘌呤酸通过酯健结合即构成核苷酸或者脱氧核苷酸.更核苷酸(5c-AMP)和3c-胞嘧啶脱氧核苷酸(3c-dC-确切地讲,一个核苷分子或一个脱氧核苷分子中的MP).这里要说明的是:在戊糖上的所有的游离羟基戊糖C5c或C3c原子上的羟基与一个磷酸分子结合,(如核糖上的第2c位、第3c位及第5c位;脱氧核糖上就形成一个核苷酸(或脱氧核苷酸),有时也称为磷的第3c位及第5c位)都可以与磷酸发生酯化反应,酸核苷.如一磷酸腺苷(AMP)和一磷酸脱氧腺苷但生物体内多数核苷酸都是5c核苷酸.一般代号可(dAMP),这里/d0表示脱氧(deoxy-)之意.磷酸与略去5c,如5c-腺嘌呤核苷酸简称腺苷酸,其它类推.\n6期许进等:DNA计算机原理、进展及难点(Ⅲ):分子生物计算中的数据结构与特性873图4两种核苷酸212核糖核酸和脱氧核糖核酸是由Watson和Crick在1953年提出来的双螺旋结我们把含有一个磷酸基的核苷酸称为一磷酸核构模型(如图5(b)所示).其主要结构特点如下:苷.多个核苷酸以磷酸与戊糖相连顺序而成长链的(1)DNA分子是两条反向平行的多聚脱氧核多核苷酸分子,即成核酸的基本结构.所以,核酸的苷酸链,围绕着同一中心轴盘旋而形成右手双螺旋一级结构是指:核酸分子中核苷酸的排序(又称碱基结构;顺序)以及核苷酸之间的连接方式.DNA分子的多(2)每条主链由磷酸和脱氧核糖相间连接而核苷酸链是以数量不等的4种脱氧核苷酸通过3c~成,位于螺旋外侧,碱基位于螺旋的内侧.碱基平面5c磷酸二酯键连接起来.所以,DNA分子的主链骨与螺旋的中心轴垂直,螺旋表面有两条螺旋型的凹架是磷酸和脱氧核糖相间排列成的长链.线形DNA槽:大沟和小沟;分子有两个游离的末端:脱氧核糖5c-OH末端,称(3)双螺旋的直径是2nm,沿着中心轴每个螺5c-末端,以及脱氧核糖的3c-OH末端,称为3c-末旋周期有10个核苷酸对,螺距为314nm,碱对之间端.一级结构的书写方向规定为5c-末端→3c-末端,的距离为0134nm;如图5(a)所示.(4)两个键之间的碱基以氢键相互配对.A与TDNA分子的二级结构,通常又称为空间结构,配,有2个氢键;G与C配,有3个氢键,如图5所示.图5DNA分子的三种形式\n874计算机学报2007年DNA分子的三级结构是指双螺旋DNA分子故通过把核苷酸序列简称为碱基序列.并通常用A,的扭曲或再螺旋.超螺旋是DNA三级结构的一种C,G和T这四个字母来表示.如图5(a)中的核苷酸重要的存在形式,如图5(c)所示.一般细胞线粒体序列可表示为内的DNA大多以这种形式存在.5cACTG3c.碱基配对是DNA分子很重要的一个性质,无在DNA计算中,已有不少学者以单链DNA分论是在自然界还是基于人工技术的基因工程等,特子作为信息处理的数据.如Adleman在文献[7]中别是在本文所言的DNA计算机,这个性质是最基所建立的求解有向图的Hamiltion有向路DNA计本最重要的.故我们在图6中给出了碱基配对之间算模型,利用单链DNA分子表示图的顶点,而构造的示意图.DNA分子的双链通过碱基配对形成氢键图的边时则以边相邻的两个顶点的各一半序列构进而形成双螺旋结构.因为几何形状等原因,A只与成.通过边的补链为模板进行杂交反应来获得所需T配对,G只与C配对.A与T之间可形成2个氢要的有向路径.键,G与C之间可形成3个氢键.配对的碱基在同以单链DNA分子为数据的DNA计算模型已一个平面上.经不少,可参见文献[8].312双链DNA分子在DNA计算中,有些模型是以双链DNA分子作为数据直接进行编码的.其实,对于DNA计算而言,双链与单链编码没有本质性区别,原因是单链的DNA分子在整个DNA计算中一般通过杂交反应变成双链.这是其一;其二,知道单链DNA分子,通过Waston-Crick互补关系实际上就等于知道了该单链DNA分子所对应的双链DNA分子,反过来,知道了一个双链DNA分子,同样通过Waston-Crick互补关系,自然等于知道了单链DNA分子.所以,在DNA计算中,应根据具体问题,可灵活采用单链还是双链DNA分子.这里,我们只给出本文对双链DNA分子的一些记号.对于一个给定的双链DNA分子,如5cACTGTTAAGA3c3cTGACAATTCT5c我们通常用英文大写字母来表示,如用X来表示上述双链DNA分子,即图6DNA双链分子中的碱基配对5cACTGTTAAGA3cX=3cTGACAATTCT5c3DNA计算中所用到DNA分子的类型我们通常用对应的英文小写字母来表示双链DNA分子中5c→3c的单链DNA分子,即双链的上半部在DNA计算研究中,几乎涉及到所有的DNA分,而用小写英文字母来表示双链DNA分子中分子类型,诸如单链DNA分子、双链DNA分子、发3c→5c的单链DNA分子,即双链的下半部分.于-卡DNA分子、环状DNA分子等.是,对于上例有:x=5cACTGTTAAGA3c;x=311单链DNA分子3cTGACAATTCT5c.所谓单链DNA分子,实际上就是DNA分子的有时在不致混淆的情况下,略去5c-端和3c-端.一级结构,即不同的核苷酸之间通过磷酸二酯键连如对上例有x=ACTGTTAAGA.我们把x称为X--接起来的一个核苷酸序列.由上一节已经知道,的上链,而把x称为X的下链.把x与x称为互补DNA分子的一级结构中磷酸与脱氧核糖的结构是链.通常将双链DNA分子X简记为不变的,而只有不同的核苷酸中的碱基序列在变化,X=R(x),\n6期许进等:DNA计算机原理、进展及难点(Ⅲ):分子生物计算中的数据结构与特性875[19]即表示由单链DNA分子x可生成双链DNA分子X.的研究组首先给出的.进一步的工作见文献[20].313发夹DNA分子314质粒DNA分子一些长的单链DNA分子在一定的条件下,通质粒是基因工程中常用的载体之一,它具备作过氢键引力,按照Waston-Crick碱基配对原则,使为基因工程的载体所具备的条件.质粒是一种亚细得A与T、G与C之间形成氢键,从而导致部分单胞有机体,没有蛋白质外壳,没有细胞外的生命周链DNA分子之间通过杂交形成双链,而部分没有期,它只能在寄主细胞内独立地增殖,并随着寄主细杂交.例如,对于单链DNA分子胞的分裂而被遗传下去,但它离开寄主的细胞就不x=5cACTGTTAAGAGGGGATTATTCTTAACAGT3c能存活,它控制的许多生物学功能也是对寄主细胞容易看出前10个与后10个碱基序列对应Waston-的补偿.质粒具有自主复制和转录能力,能使子代细Crick匹配,且在一定的条件下形成的结构如图7胞保持它们恒定的拷贝数,可表达它携带的遗传信所示.息.质粒可独立地游离在细胞质内,也可以整合到细菌染色体中.业已发现,质粒分子存在于诸如原核生物细胞、真核生物细胞、格兰氏阳性细菌、格兰氏阴性细菌、真菌的线粒体、大肠杆菌等细胞和菌体中.图7一个发夹DNA分子结构质粒是一种染色体外的稳定遗传因子,大小在1~200kb,具有双链闭合环状结构的DNA分子,质我们把如图7所示的这种一端是双链,另一端粒的结构是比较简单的,它比病毒的结构还要简单.是环状的DNA分子称为发夹DNA分子构型,或简在大肠杆菌的菌株中,科学家们已经寻找到了称为发夹DNA分子.我们把发夹DNA分子中的双多种不同类型的质粒,其中比较熟悉的有三种:F质链部分称为它的径,把环状部分称为环.这方面进一粒、R质粒和Col质粒.其中的F质粒(又称F因子,步的讨论见第415节.或者性质粒)能够使寄主染色体上的基因和它们一发夹DNA分子在DNA计算与DNA计算机道转移到原先不存在该质粒的受体细胞中去;R质的研究中占有很重要的地位:(1)利用发夹DNA分粒通称抗药性因子,它们的编码有一种或多种抗菌子制作成分子信标,分子信标的基本结构如图8所素抗性基因,并且在通常能够将此种抗性转移到缺示.分子信标技术在DNA计算中主要用于解的检乏该质粒的适宜的受体细胞,使得该受体细胞也获测.这方面的研究已经不少,有关这方面更深入的讨得同样的抗菌素抗性能力;Col质粒,即所谓的产生论可参见本文中后面的有关章节;(2)将发夹DNA大肠杆菌素因子.它们编码有控制大肠杆菌素合成分子直接用于DNA计算,如文献[28]中利用发夹的基因.大肠杆菌素是一类可以使不带有Col质粒DNA分子建立了用于求解可满足性问题的DNA的亲缘关系密切的细菌菌株致死的蛋白质.计算模型;(3)将发夹DNA分子应用于疾病诊、疗细菌质粒有线性质粒、杀伤质粒和环形双链质DNA计算机模型的研究,其中在径部为诊断部分,粒DNA.目前发现的质粒的绝大多数是后者.环形而环部则为抑制疾病发展的一段DNA序列.这方双链的DNA分子具有三种不同的构型:共价闭合面开拓性的工作是以色列学者EhudShapiro领导环形DNA(SC构型,cccDNA)、开环DNA(通常称[21]为OC构型,即ocDNA)以及线性L构型.现简述如下:①共价闭合环形,是指两条多核苷酸链都保持完整的环形结构,这样的DNA通常呈现超螺旋的SC型;②开环,是指另一条链出现有至少一个缺口;③线性,是指当质粒DNA分子经过适当的核酸内切限制酶切开之后,发生双链断裂而形成线性分子.图8分子信标的工作原理示意图由于目前DNA计算中主要采用环状双链的质\n876计算机学报2007年粒DNA分子,故下面介绍一下环状双链质粒DNA315具有粘性末端的DNA分子分子的基本特性.我们把形如图10中所示的DNA分子称为具质粒DNA的编码中,适用于作为基因克隆载有粘性末端的DNA分子.显然,在DNA计算中,这体的所有质粒DNA分子,都必定包含下面三种共种类型的DNA分子是人工合成的.作为信息处理同的组成部分,即复制子结构(replicon)、选择性记的数据,这种类型的分子在DNA计算中具有广泛号和克隆位点.其中,复制子结构包括一个复制起始的应用.如应用于所谓的粘贴DNA计算模型.应用位点(origin,简称ori),控制复制频率的调控基因以于图顶点着色DNA计算机模型,更一般地讲,几乎及一些复制子编码基因.对这里的克隆位点,我们主可应用于图论中的所有问题的研究之中.在文献要强调MCS(多克隆位点),包含若干单一限制性酶[25-27]中,我们可以看到关于具有粘性末端DNA切位点,可供外源DNA定点插入.图9中给出了一分子在DNA计算中更为深入的应用.个质粒DNA分子的基本结构.在质粒DNA分子的基本结构中,我们对应于外源核苷酸序列的插入和[21]删除采用的基本上是Ⅱ型核酸内切限制酶.Ⅱ型核酸内切限制酶只有一种多肽,并通常以同源二聚图10具有粘性末端的DNA分子类型体形式存在,其特点是:(i)在DNA分子双链的特异性识别序列部位,切割DNA分子产生链的断裂;(ii)2个单链断裂部位在DNA分子上的分布通常4DNA分子的某些基本性质不是彼此直接相对的;(iii)断裂的结果形成的DNA片断,也往往具有互补的单链延伸末端.本节将与DNA计算有关的DNA分子中的一些性质介绍给读者.这些性质在DNA计算研究中是基本的,是作为DNA计算乃至一般基因工程学者应该掌握的.本节的内容是为非生物学专业的学者而引入的,但这些内容是很不够的,在对DNA计算进行深入研究时需进一步掌握有关DNA分子的特性与结果.411DNA分子的变性和复性核酸的变性是指核酸分子中双螺旋区内的碱基对之间的氢键受到某些物理的,或者化学的因素作用而断开,变成单链的过程.变性后的核酸将失去其部分或全部的生物活性.需要注意的是:核酸在变性过程中仅仅是碱基之间氢键的断开,而磷酸二酯键并未受影响,所以它的一级结构保持不变.更确切图9质粒DNA分子结构图示地讲,双链DNA分子(特别是DNA分子的双螺旋)正是由于质粒DNA分子的上述特性,才使得结构的氢键能够在诸如温度升高,介质PH值<4DNA计算得以实施.其质粒DNA计算模型是充分或>10,变性剂(如有机试剂甲醇、乙醇、尿素及甲酰利用质粒DNA分子上的基因位,通过核酸内切酶胺等)能够发生断裂,当所有的氢键都被破坏时,双与连接酶的作用,可产生两种状态,分别用0和1来链DNA分子多核苷酸链就完全分开,这一过程就表示这两种状态,这样,每个质粒体与传统计算机中称为DNA分子的变性或解链.k-位数据寄存器作用相同.这就是质粒DNA计算一般变性分为两类:由温度的升高而引起的变的基本思想.性称为热变性;由酸碱度的改变而引起的变性称为Head等人在1999年首先建立了基于质粒酸碱变性.变性过程实际上只发生在一个较狭窄的DNA分子的、用于求解图的顶点的最大独立集问题温度范围,同时伴有物理性状的改变,其中最有意义[22]的质粒DNA计算模型,其后在这方面进一步的的是吸光度的改变.双链DNA的吸光度(A260)比同研究见文献[23-24].量单链DNA碱基的吸光度要低,所以可用吸光度\n6期许进等:DNA计算机原理、进展及难点(Ⅲ):分子生物计算中的数据结构与特性877的增加来表示DNA的变性过程(如图11所示).412解链温度Tm值解链温度Tm,定义为使得双链DNA分子在变性过程中,有50%的碱基对变成单链时的温度.它是评价DNA分子的热力学稳定性的另一个重要的参数.一个DNA分子的Tm值不仅与浓度、溶液的PH值有关,而且与其分子大小及所含碱基中的GC含量有关,并且与碱基序列的排列有关.由于解链温度Tm在DNA计算中占有很重要的位置,故我们在此专门作为一个小节给予介绍.DNA计算中的一个主要的生化操作是双链DNA分子的解链问题.由于一般参与生化反应的图11吸光率与温度的关系示意图DNA分子是巨量的,并要求在一个很短的时间段内所谓复性(renaturation)是变性过程的逆过程,对所需要的DNA分子全部进行解链,即就是要求即对于变性后的二条完全互补的单链DNA分子,作为数据的DNA分子具有尽可能相同或者相近的在适当的条件下恢复到双链、甚至天然双螺旋结构解链温度Tm.的过程.热变性的DNA分子一般经过冷却后即可为了达到这个目的,首先,我们在设计DNA序复性,因此,此过程有时也称退火(annealing).关于列时必须要求所有的双链DNA分子具有相同的氢DNA分子的变性与复性的过程示意图见图12.复键数目,原因是DNA分子双链的解链实际上就是性温度一般比该DNA分子的解链温度Tm值(此概将双链中的氢键打开;其次,考虑到碱基堆积力的影念下小节中给予介绍)低25e.响,我们在设计DNA序列时,还应该考虑DNA序列的次序,即如何针对DNA计算问题,设计DNA序列来控制解链温度Tm值,以保持所有作为数据的DNA分子的解链温度尽可能相近.在考虑DNA序列次序的研究方面,其方法已经不少,我们将在另文较为详细地讨论.413双链DNA分子间的作用力上面我们已经讲过,DNA分子能够形成双链的重要原因是氢键之间的引力,更确切地讲,是指碱基A与T、碱基之间的氢键作用力.由于G与C之间存在3个氢键,而A与T之间存在2个氢键,故G图12DNA分子的变性和复性与C之间的引力大于A与T之间的引力.因而,同复性后的DNA在某些理化性质及生物活性也样等长度的双链DNA分子,由于GC含量的不同导可得到部分和全部的恢复.复性过程中,紫外吸收值致双链的稳定性不同,GC含量越多,稳定性越好;降低,此现象称为减色效应.复性反映与许多因素有GC含量越少,稳定性越差.DNA分子内部中的另关,其主要的如下:外一个主要的作用力是碱基之间的堆积力.由于这(1)复性时的降温速度必须缓慢,否则,复性过两种主要力的作用,给DNA计算带来一些麻烦与程不可能完成.我们把从高温缓慢冷却使DNA变困难:其一、单链DNA分子在一定的温度与环境下性的过程称为退火;而把从高温迅速冷却至低温容易形成发夹分子构型,给特异性杂交带来困难;其(4e以下)的过程称为淬火.淬火后的DNA不能二、在设计作为信息处理的数据DNA分子的编码复性.时至少带来两个困难的约束条件.(2)DNA的浓度越大,互补的DNA片段碰撞414DNA分子的复制的机会越多,当然易于变性;DNA分子可以进行复制,在DNA聚合酶的作(3)DNA片段越长,DNA分子的排列越复杂,用下,一个DNA分子可复制成两个在结构上完全互补碱基相遇的机会也越小,复性越困难.相同的DNA分子.在DNA分子进行自我复制时,\n878计算机学报2007年复制的两条互补链分开,然后在每条链上按碱基配完全对接上,形成原来的双链DNA分子的过程.但对规律形成互补的新链,以组成新的DNA分子.每是,所谓的DNA分子的杂交则是将来源不同的个DNA分子的二条链都可以作为形成互补链的模DNA片段,按碱基互补关系形成杂交双链分子或者板.复制的结果是:形成了与原来DNA分子完全相部分双链的过程.杂交的类型主要有如下几种:同的2个子代DNA分子,如图13所示.(1)完全性杂交所谓完全性杂交,是指按照Watson-Crick互补原则完全互补的两条DNA序列之间在一定的条件下相互匹配的碱基之间均形成氢键的现象称为完全性杂交,如图14(a)所示.可以说,DNA计算的核心工作就是围绕着进行特异性的完全杂交展开的一系列研究工作.(2)假阳性杂交所谓假阳性杂交,是指不完全互补的DNA分子在适当的条件下也能够杂交形成双链分子的现图13DNA自复制示意图象,如图14(b)、(e)所示.假阳性主要是由于杂交的2个DNA分子间的序列有足够的/相似度0造成的.415DNA分子间的杂交DNA计算中的信息处理手段就是DNA分子这种杂交现象在DNA计算中一般是要想方设法回间的所谓的/特异性杂交0.DNA计算实际上就是针避的现象,然而,在DNA计算乃至整个过程中会经对DNA分子的间的特异性杂交展开一系列的工常出现.为了克服这种现象,首要的问题是在DNA作,诸如编码设计问题、解空间的设计问题、解的检序列的编码问题上设法解决;其次考虑在实验条件测问题等.正因为DNA分子在DNA计算中的关键上进行处理.而重要的作用,我们在这一小节里专门就DNA分(3)假阴性杂交子杂交的有关概念、基本性质等给予介绍.所谓假阴性杂交,是指完全互补的DNA分子在411节中我们讨论了DNA分子的变性与复在反应过程中由于种种原因而没有完全杂交的现性问题.DNA分子的变形使得双链DNA分子间的象,如图14(c)所示就是其中的一种.我们把这个具氢键断开,而复性则恰恰相反,是将一对被断开的哪体的现象也称为位移杂交.假阴性现象出现的主要两条互补的单链DNA分子在一定的温度、一定的原因是由反应条件及生化操作本身的失误引起的.环境等条件下通过退火等手段使得它们对应的氢键所以,我们在进行生化实验时需要精心操作.图14DNA分子杂交的类型(4)发卡构形杂交有时候反而可利用这种杂交来帮助DNA计算进行所谓发卡构形杂交,是指一个单链的DNA序一些信息处理.如文献[28]中所建立的用于求解可列自身的碱基序列之间在一定的条件下通过氢键引满足性问题的DNA计算模型时,使问题的非解形力形成氢键的现象.如图14(d)所示.这种杂交现象成发卡构形,并在发卡构形上加上一个酶切位点,将一般情况下当然是我们不希望产生的.我们通过生其切除.化操作控制,对发卡构形杂交现象是容易克服的.但杂交双链可以在DNA与DNA链之间,也可在\n6期许进等:DNA计算机原理、进展及难点(Ⅲ):分子生物计算中的数据结构与特性879RNA与DNA链之间以及PNA与DNA之间形成.modelsofDNAcomputer.ChineseJournalofComputers,2007,30(6):881-893(inChinese)由于这两种杂交现象在目前的DNA计算,或者(许进,谭钢军,范月科,郭养安.DNA计算机:原理、进展及RNA计算,或者PNA计算中尚未使用,故这里不难点(Ⅳ):论DNA计算模型.计算机学报,2007,30(6):再讨论.881-893)[9]BaumEB.DNAsequencesusefulforcomputation//Proceed-5结束语ingsofthe2ndDIMACSWorkshoponDNABasedComput-ers.PrincetonUniversity,1996:235-241[10]Feldkampetal.ADNAsequencecompile//Proceedingsof本系列文章主要讨论了DNA计算机理论中的the6thDIMACSWorkshoponDNABasedComputers,2000[29-30]几个基本理论问题.本文是系列文章中的第三[11]GarzonM,DeatonR,NinoLF,StevensSE,WittnerM.篇,讨论DNA计算机的数据库.重点讨论了作为GenomeencodingforDNAcomputing//ProceedingsoftheDNA计算机/数据0的DNA分子的结构、特性与功3rdDIMACSWorkshoponDNA-BasedComputing.Univer-能,并指出了不同DNA分子在DNA计算中的作sityofPennsylvania,1997:230-237用,在DNA计算中的重要性.目前DNA计算机的[12]DeatonRetal.ADNAbasedimplementationofanevolu-tionarycomputation//ProceedingsIEEEConferenceonEvo-研究正处在一个关键的转折阶段,即如何摆脱解空lutionaryComputation.Indiana,1997:267-271间的/枚举0思想,而应该建立将优化计算与生物操[13]FrutosAGetal.Demonstrationofaworddesignstrategy作有机结合起来的思路.这种思想就自然而然地摆forDNAcomputingonsurface.NucleicAcidsResearch,脱了/枚举0思想的困惑,即消除DNA计算机研究1997,25(23):4748-4757中的最大障碍)))解空间指数爆炸问题.为此,熟练[14]HartmeminkAJ,GiffordDk,KhodorJ.Automatedcon-掌握核酸结构与特性以及有关生物酶与生化操作是straint-basedsequenceselectionforDNAcomputation.Bio-System,1999,52:227-235必须的,特别,对于一个从事数学、物理,或者电子计[15]DeatonR,FranceschettiDR,GarzonM,RoseJA,Murphy算机学者,或者信息科学的科技工作者,更应掌握生RC,StevensJrSE.Informationtransferthroughhybridiza-化实验等方面的知识.这也是本文的目的.有关这方tionreactionsinDNAbasedcomputing//GeneticProgram-面更深入的知识可参考文献[21,31]等.ming1997:ProceedingsoftheSecondAnnualConference,AAAI.StanfordUniversity,1997:463-471[16]TanakaFumiaki,KamedaAtsushietal.Designofnucleic参考文献acidsequencesforDNAcomputingbasedonathermodynamicapproach.NucleicAcidsResearch,2005,33(3):903-911[1]ConradM.Ondesignprinciplesforamolecularcomputer.[17]LiuX-iKui.ThestudiesonDNAoptimalencodingandgenet-CommunicationoftheACM,1985,28(5):464-480icalgorithmwithapplications[Ph.D.dissertation].Depart-[2]BirgeRP.Protein-basedcomputers.ScientificAmerican,mentofControlScienceandEngineering,HuazhongUniver-1995,272(3):90-95sityofScienceandTechnology,Wuhan,2003(inChinese)[3]FreemantleM.Proteindevicesmayincreasecomputerspeed(刘西奎.DNA编码优化与遗传算法应用研究[博士学位论andmemory.ChemicalandEngineeringNews,May22,文].华中科技大学控制科学与工程系,武汉,2003)1995:10-11[4]LandweberLF.RNAbasedcomputing:someexamples[18]ZhangKai,PanLin-Qiang,XuJin.AglobalheuristicallyfromRNAcatalysisandRNAediting//LandweberLF,searchalgorithmforDNAencoding//MasamiHagiyaed.Pre-proceedingsofBIC-TA2006.Wuhan,China,2006:BaumEBeds.DNABasedComputersⅡ:Proceedingsofa20-29DIMACSWorkshop1996.AmericanMathematicalSociety,1996:181-189[19]YaakovBenenson,TamarPaz-Elizur,RivkaAdar,Ehud[5]CukrasA,FaulhammerD,LiptonRetal.Chessgames:AKeinan,ZviLivneh,EhudShapiro.Programmableandau-modelforRNA-basedcomputation.BioSystems,1998,52:tonomouscomputingmachinemadeofbiomolecules.Nature,35-452001,414(22):430-434[6]FaulhammerDirk,CukrasAnthonyR,LiptonRichardJ[20]YaakovBenenson,BinyaminGil,UriBen-Dor,RivkaAdar,etal.Molecularcomputation:RNAsolutionstochessprob-EhudShapiro.Anautonomousmolecularcomputerforlog-ilems.Biochemistry,2000,97(4):1385-1389calcontrolofgeneexpression.Nature,2004,429:423-429[7]AdlemanL.Molecularcomputationofsolutiontocombinato-[21]WuNa-iHu.GeneEngineeringPrinciple.Beijing:Sciencerialproblems.Science,1994,66(11):1021-1024Press,2002(inChinese)[8]XuJin,TanGang-Jun,FanYue-Ke,GuoYang-An.DNA(吴乃虎编著.基因工程原理(第二版).北京:科学出版社,computerprinciple,advancesanddifficulties(Ⅳ):Onthe2002)\n880计算机学报2007年[22]HeadT,RozenbergG,BladergroenRB,BreekCKD,tions.ChineseScienceBulletin,2004,49(8):1-9LommersePHM,SpainkHP.ComputingwithDNAby[28]SakamotoK,GouzuH,KomiyaKetal.Molecularcomputa-operatingonplasmids.BioSystems,2000,57:87-93tionbyDNAhairpinformation.Science,2000,288(49):[23]ZhangLian-Zhen,LiuGuang-Wu,XuJin.Theresearchof1223-1226DNAcomputingmodelbyoperatingonplasmids.Computer[29]XuJin,ZhangLei.DNAcomputerprinciple,advancesandEngineeringandApplications,2004,(4):51-53(inChinese)difficulties(I):Biologicalcomputingsystemanditsapplica-(张连珍,刘光武,许进.基于质粒的DNA计算模型研究.计tionstographtheory.ChineseJournalofComputers,2003,算机工程与应用,2004,(4):51-53)26(1):1-11(inChinese)[24]ZhangLian-Zhen.TheapplicationsofDNAcomputingmodel(许进,张雷.DNA计算机原理、进展与难点(I):生物计算系byoperatingonplasmidstographandoptimizationproblems统及其在图论中的应用.计算机学报,2003,26(1):1-11)[M.S.dissertation].DepartmentofControlScienceandEn-[30]XuJin,HuangBu-Yi.DNAcomputerprinciple,advancesgineering,HuazhongUniversityofScienceandTechnology,anddifficulties(Ⅱ):SettingupthedatabaseofDNAcom-Wuhan,2003(inChinese)puterbythesynthesisofDNAmolecules.ChineseJournalof(张连珍.质粒DNA计算模型在图与组合优化中某些问题的Computers,2003,28(10):1583-1591(inChinese)应用[硕士学位论文].华中科技大学,武汉,2003)(许进,黄布毅.DNA计算机:原理、进展及难点(Ⅱ):计算机[25]RoweisS,WinfreeE,BurgoyneR,Chelyapov,GoodmanM,/数据库0的形成)))DNA分子的合成问题.计算机学报,RothemundP,AdlemanLA.StickerbasedmodelforDNA2005,28(10):1-11)computation//Proceedingsofthe2ndDIMACSWorkshopon[31]JosephSambrooketal.MolecularCloning:LaboratoryMan-DNABasedComputers.Princeton,1999,44:1-29ual.ColdSpringHarborLaboratory,2000(inChinese)[26]XuJinetal.StickerDNAcomputermodel(I):Theory.(J.萨姆布鲁,D.W.拉赛尔著.黄培堂译.克分子克隆实验ChineseScienceBulletin,2004,49(7):1-8指南(第三版).北京:科学出版社,2002)[27]XuJinetal.StickerDNAcomputermodel(II):Applica-XUJin,bornin1959,professor,ZHANGShe-Min,bornin1962,Ph.D.candidate,pro-Ph.D.supervisor.Hisresearchinter-fessor.Hisresearchinterestincludebio-computerandgraphestsincludebio-computer,graphtheorytheory.andsystemengineering,bio-informat-FANYue-Ke,bornin1959,associateprofessor.Hisics,neuralnetworks,geneticalgorithmsresearchinterestincludeDNAcomputingandgraphtheory.etc.GUOYang-An,bornin1960,associateprofessor.Hisresearchinterestingincludescomputerscienceandmanage-mentscience.BackgroundThisresearchissupportedbytheNationalNaturalSc-iquences,synthesizingDNAmolecules,settingupthemodel,enceFoundationofChina(60533010,30670540),andthedetectingsolutions,etc.Theresearchgrouphasbeenwork-NationalHighTechnologyResearchandDevelopmentPro-ingonmanyaspectsofDNAcomputingsince1996.Theygram(863Program)ofChina(2006AA01Z104):Researchhavepublishedamonographandmorethan100papersonontheTheory,ModelandMethodofDNAComputeretc.DNAcomputingandDNAcomputer.Inthispaper,theau-TheprojectsmainlyfocusonDNAcomputermodelsforpro-thorspresenttheStructureandCharacterof"Data"inDNAcessinggraphicalmessages,includingencodingDNAse-Computing.ThisisabasictheoryinDNAcomputer.